Bioinformatische Genomforschung
Bislang gibt es kein räumlich-funktionales Modell des menschlichen Genoms. Wir wissen nicht, welche genetischen Varianten welche Effekte auf die dreidimensionale Anordnung und damit verbunden die Regulation der Genexpression haben. Wir suchen mit Hilfe bioinformatischer Methoden anhand eigener und öffentlicher Daten nach Auffälligkeiten, um den Einfluss der Telomere auf die Expression von Genen zu charakterisieren.
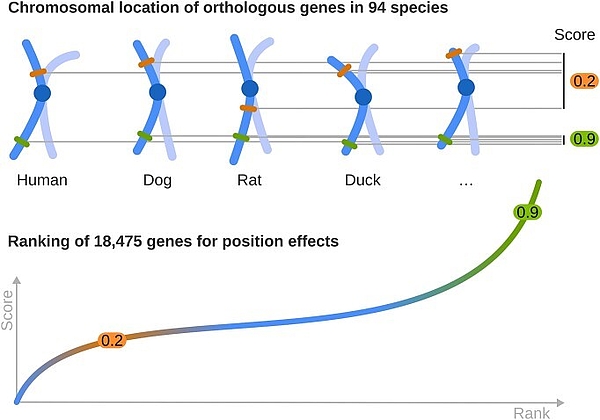
Es werden nicht alle Gene in gleicher Weise von Telomeren kontrolliert. Unsere Hypothese ist, dass Gene, für die die relative Lage zu den Telomeren besonders wichtig ist, Spezies-übergreifend in konstantem Abstand zu den Telomeren kodiert sind. In einer medizinischen Doktorarbeit wurde hierzu ein Web-Interface etabliert (https://tpe-old.uni-rostock.de/; Projahn et al., 2024). Die daraus abgeleiteten Hinweise konnten wir im Labor dazu nutzen, das Tumor-Suppressor-Gen PPP2R2C als TPE (Telomer-Positions-Effekt)-Gen zu identifizieren. Dies bedeutet, dass die Regulation der Genexpression direkt von der Telomerlänge beeinflusst wird (Jäger et al., 2022).
TELOMERLÄNGEN-BESTIMMUNG DURCH SEQUENZIEREN
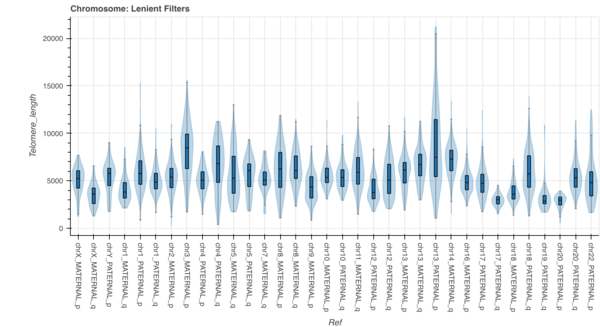
Die exakte Bestimmung von Telomerlängen durch Sequenzierung war bislang nicht möglich, bedingt durch Nachteile der verfügbaren Sequenziertechniken (Sequenzierung von zumeist nur kurzen Fragmenten von bis zu 600 Basenpaaren) sowie die repetitive Sequenz und fragile Struktur der Telomere.
Wir haben, gefördert durch ein FORUN Projekt der Universitätsmedizin, ein erst jüngst beschriebenes Verfahren (Schmidt et al., Nat. Commun. 2024) zur Chromosomen-spezifischen Messung der Telomerlänge etabliert. Dieses Verfahren basiert auf der sehr große Leselängen erlaubende Nanopore-Sequenziertechnologie. Hierdurch können erstmals abnorme Telomerstrukturen bei seltenen Erkrankungen charakterisiert und die exakten Telomerlängen Chromosomen-spezifisch bestimmt werden. Anhand der Sequenz-Information lassen sich Veränderungen auch in der Familie verfolgen, also den Einfluss des Vaters oder der Mutter auf die Telomerlängen beschreiben.
Referenzen:
1.) Projahn EF, Fuellen G, Walter M, Möller S. Proposing candidate genes under telomeric control based on cross-species position data. NAR Genom Bioinform. 2024;6:lqae037.
2.) Jäger K, Mensch J, Grimmig ME, Neuner B, Gorzelniak K, Türkmen S, Demuth I, Hartmann A, Hartmann C, Wittig F, Sporbert A, Hermann A, Fuellen G, Möller S, Walter M. A conserved long-distance telomeric silencing mechanism suppresses mTOR signaling in aging human fibroblasts. Science Advances 2022;8:eabk2814.
3.) Möller S, Saul N, Projahn EF, Barrantes I, Gézsi A, Walter M, Antal P, Fuellen G. Gene co-expression analyses of health(span) across multiple species. NAR Genom Bioinform. 2022;4:lqac083.
4.) Schmidt TT, Tyer C, Rughani P et al. High resolution long-read telomere sequencing reveals dynamic mechanisms in agin g and cancer. Nat Commun 2014;15:5149.
Beteiligte Wissenschaftler:


